Heiliger Gral der Zuckerbiologie
ETH-Biologen haben die Struktur eines Bakterien-Enzyms aufgeklärt, das Proteine mit Zuckern verknüpft. Die Erkenntnisse über die Struktur ermöglichen Einblicke in den Mechanismus dieser Modifikationen. Zudem dürften sie die Entwicklung neuer Methoden zur Herstellung therapeutisch nutzbarer «süsser Proteine» auslösen.
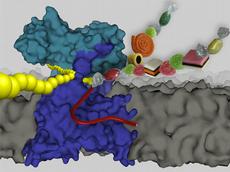
Das Enzym, dessen Struktur die ETH-Biologen soeben bestimmt haben, trägt einen fast unaussprechlichen Namen: Oligosaccharyltransferase. Dieses Protein sitzt in Membranen von gewissen Bakterien, den Gram-negativen, zu denen einige Krankheitserreger wie verschiedene Campylobacter-Arten gehören.
Diese Bakterien haben dank dieses Enzyms eine besondere Fähigkeit, die ansonsten nur eukaryotische Zellen, wie Hefen oder Säugerzellen, haben: Sie können verschiedenste Proteine mit Zuckern verknüpfen, sogenannt glykosylieren. Diese Glykosylierung findet an einer bestimmten Stelle des Proteins statt, und zwar an einem Protein-Baustein namens Asparagin. An diesen Baustein kann das Enzym über eine Stickstoffgruppe Zucker binden, weshalb der Vorgang auch N-Glykosylierung heisst.
Strukturaufklärung geht vor
Wie das genau vor sich geht, darüber rätselten die Biologen – und auch Chemiker – lange. Denn diese Reaktion ist träge, zu wenig reaktiv ist der Stickstoff-Rest am Asparagin. «Der Knackpunkt war, wie Asparagin aktiviert wird, um mit dem Zucker eine Verbindung einzugehen», sagt Christian Lizak, Doktorand im Institut für Mikrobiologie der ETH Zürich.
Um den chemisch schwer nachvollziehbaren Vorgang zu verstehen, war es deshalb nötig, erst die genaue Struktur des Enzyms zu kennen. Die ETH-Forscher haben darum «PglB» aus dem Bakterium Campylobacter lari isoliert und kristallisiert. «Dieser Schritt war besonders anspruchsvoll, die Arbeiten dazu haben vor acht Jahren begonnen», sagt Lizak, der Erstautor der Studie ist, die soeben in der Fachzeitschrift Nature veröffentlicht wurde.
Die Kristalle wurden an einer hochauflösenden Strahlenquelle der Swiss Light Source (SLS) am Paul Scherrer Institut mit Röntgenstrahlen beschossen. Aus den Röntgenbildern berechneten die Forscher schliesslich jede einzelne Atomposition mit einer Auflösung von 3,4 Angström, was einer Grössenordnung von wenigen milliardstel Metern entspricht.
Lebenswichtige Funktion
In Bakterien ist das Enzym «PglB» für die Glykosylierung verantwortlich. Diese Oligosaccharyltransferase ist im Vergleich zum Enzym in tierischen Zellen, die aus acht verschiedenen Untereinheiten besteht, relativ einfach aufgebaut. Die zentrale Einheit des tierischen Enzyms ist jedoch dem «PglB»-Protein von Bakterien sehr ähnlich.
Mag die N-Glykolysierung für Bakterien eine nicht lebenswichtige Funktion sein, so können Zellen höherer Lebewesen ab Stufe Hefe bis hin zu Säugern ohne diese nicht leben. Schaltet man bei Hefezellen diese Maschinerie ab, so sterben die Zellen innert kurzer Zeit. Denn die N-Glykosylierung ist für eine Vielzahl von Lebensfunktionen notwendig. Mehr als die Hälfte aller Proteine in einer eukaryotischen Zelle werden nach ihrer Synthese mit Zuckern «versüsst».
«Die Struktur der Oligosaccharyltransferase ist so etwas wie der «heilige Gral» der Zuckerbiologie», sagt Lizak. Mehrere weltweit führende Forscherteams aus Oxford, des Caltechs oder des MIT hätten bis anhin vergeblich versucht, diese Struktur zu lösen. Den ETH-Forschern ist es nun gelungen, einen grossen Schritt vorwärts zu machen. «Die Grundlage unseres Erfolgs war die Kollaboration von unterschiedlichen Kompetenzzentren innerhalb der ETH», betont der Doktorand.
Das ursprüngliche System der bakteriellen N-Glykosylierung stammt aus dem Labor von ETH-Professor Markus Aebi. Lizak hat dort seine Doktorarbeit gemacht und die Geheimnisse der Zuckerbiologie erlernt. Als er bei der Kristallisation des Enzyms an seine Grenzen stiess, konnte er während seiner Dissertation in die Gruppe von Kaspar Locher wechseln. Locher ist Experte für die Reinigung, Kristallisation und Strukturaufklärung von Membranproteinen, und zu dieser Proteingruppe gehört «PglB». Dadurch gelang es, das Enzym zu kristallisieren und mit den gewonnenen Röntgendaten konnte der ETH-Professor für molekulare Membranbiologie die dreidimensionale Struktur des Proteins berechnen.
Werkzeug für Therapeutika-Herstellung
Die Enzym-Struktur liefert fundamentale Erkenntnisse zum grundsätzlichen Verständnis der N-Glykosylierung sowohl in Bakterien als auch in Eukaryoten. Der atomare Aufbau und die dreidimensionale Form zeigen auf, wie das Enzym seine Substrate erkennt, wo und wie diese an dieses andocken und wie es den verschiedenen Proteinen den Zucker «aufsetzt».
Diese Erkenntnis gibt den Forschern nun ein Werkzeug in die Hand, mit dem sie das Enzym gezielt verändern können. Das «normale» Enzym kann nämlich nur dann bestimmte Proteine glykosylieren, wenn diese eine spezifische Abfolge an Aminosäuren besitzen. Bei einem veränderten Enzym könnten aber auch andere Substrate in seine aktive Domäne passen und glykosyliert werden.
Gelingt es den Forschern, das Enzym umzunutzen, dann könnten sie es einsetzen, um in E. coli-Bakterien verzuckerte Proteine herzustellen, die beispielsweise als Therapeutika oder als Impfstoffe genutzt werden können.
«Zucker kodieren, wie DNA und Proteine, biologische Informationen und bilden die dritte Sprache des Lebens. Durch die Strukturaufklärung der Oligosaccharyltransferase wissen wir nun, wie diese Informationsträger auf Proteine gelangen können», sagt Lizak.
Literaturhinweis
Lizak C, Gerber S, Numao S, Aebi M & Locher KL. X-ray structure of a bacterial oligosaccharyltransferase. Nature online publication, 15. Juni 2011. DOI: 10.1038/nature10151








LESERKOMMENTARE