Zusammenarbeit von Proteinen neu erforscht
ETH-Forscher haben eine Methode entwickelt, mit der sie Struktur und Zusammensetzung von Proteinkomplexen erforschen können. In ihrer neusten Veröffentlichung in «Science» zeigen sie am Beispiel des Enzyms Protein-Phosphatase 2A, wie das funktioniert.
Die Protein-Phosphatase 2A (PP2A) ist ein Proteinkomplex, der aus drei Untereinheiten besteht. Sie hat in der Zelle viel zu tun: Sie entfernt Phosphatreste von verschiedensten Proteinen. Das kann deren dreidimensionale Struktur verändern sowie ihre Aktivität regulieren.
Damit PP2A seine Funktion zur richtigen Zeit am richtigen Ort in der Zelle ausführen kann, benötigt es Helferproteine, die an diesen Phosphatasekomplex binden. Auf diese Weise reguliert PP2A wie die Zelle wächst, sich differenziert, teilt, bewegt oder auf Beschädigung der DNS reagiert. Verschiedene Mechanismen steuern die Bindung dieser Helferproteine an PP2A, um deren Aktivität zielgerichtet zum Einsatzort zu geleiten und bei Dienstschluss wieder herunterzufahren.
Solche Netzwerke, in denen verschiedene Proteinkomplexe mit unterschiedlichen Funktionen zusammenarbeiten, nennt man Signalübertragungswege oder -netzwerke. Sie empfangen Signale, leiten diese weiter und lösen passende Aktivitäten der beteiligten Proteine innerhalb der Proteinkomplexe aus.
Neue Methode
Bisher war es sehr schwierig, Genaueres über die Architektur solcher Netzwerke herauszufinden. Geschweige denn über die Art und Weise, wie die beteiligten Proteinkomplexe miteinander zusammenarbeiten und Signale weitergeben - besonders bei Komplexen, die wie PP2A mit mehreren Partnern interagieren.
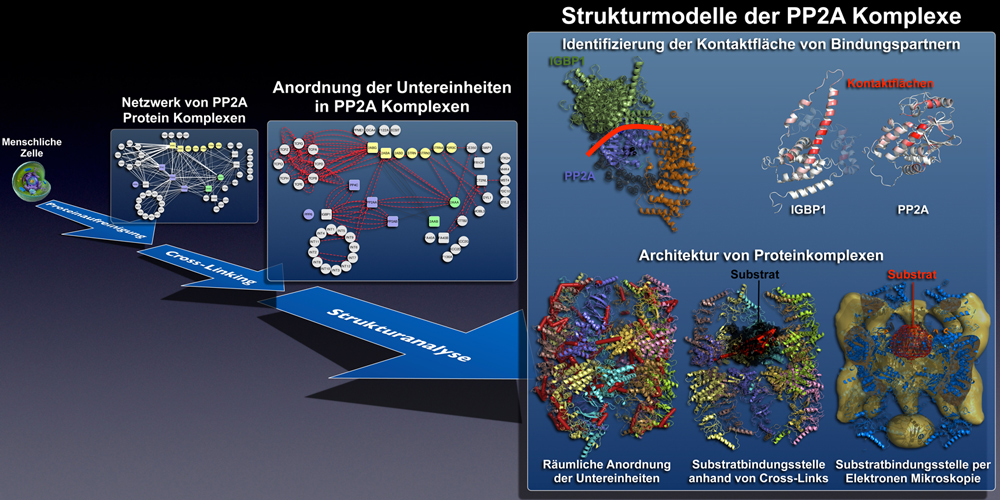
Forscher der Gruppe von Professor Ruedi Aebersold am Institut für Molekulare Systembiologie entwickelten in den letzten Jahren einen neuartigen Ansatz. «Die Methode ist eine Kombination aus chemischer Quervernetzung und Massenspektrometrie», erklärt Franz Herzog, einer der beteiligten Forscher.
Er ist der Erstautor einer neuen Publikation des Aebersold-Teams, die gerade in «Science» erschienen ist. Mit der Kombinations-Methode (abgekürzt XL-MS genannt) kann die Struktur grosser Proteinkomplexe aus mehreren Untereinheiten sowie das Zusammenspiel von Proteinkomplexen in Signalübertragungswegen auf elegante Weise aufgeklärt werden.
In ihrer Publikation beschreiben die Forscher, wie sie mit Hilfe der XL-MS-Methode die räumliche Anordnung der Untereinheiten und Helferproteine von PP2A erforscht haben. Sie konnten Fragen beantworten wie: Wie sieht die dreidimensionale Anordnung der Proteine genau aus? Wo liegen die Kontaktflächen, die die Bindung zwischen den Proteinen vermitteln? Und: Mit welchen Helferproteinen lagert sich PP2A wo und wie zusammen? Laut Herzog lassen sich mit den Resultaten sogar teilweise Aussagen über die Art der Zusammenarbeit der beteiligten Proteine machen. Etwa dann, wenn ein deaktivierendes Teilstück eines Hemmproteins ins aktive Zentrum der Phosphatase gelangt.
Entscheidende Vorteile
Mit herkömmlichen Methoden kann man in einigen Fällen zwar die Struktur einzelner grosser Proteinkomplexe sehr gut abbilden. Dafür benötigt man sie in der Regel sehr rein und in grosser Menge. Möchte man die Struktur eines Komplexes, der mehrere Untereinheiten und eine variable Zahl von Interaktionspartnern hat, aufklären, sind diese Methoden ungeeignet.
Mit der Kombination von chemischer Quervernetzung (engl. Crosslinking) und Massenspektrometrie ist das anders. Die Forscher können mit ihr Mischungen verschiedener Proteinkomplexe untersuchen. Da die XL-MS Methode sehr sensitiv ist, ist es auch möglich, Proteine in geringer Menge, die für andere Methoden nicht ausreichend angereichert und gereinigt werden können, zu untersuchen. «Dadurch eignet sich die Methode speziell gut für die Analyse heterogener menschlicher Proteinkomplexe», sagt Herzog.
Clevere Kombination
Bei der Entwicklung der XL-MS machten sich die Forscher den Umstand zunutze, dass Proteine Bindungen eingehen müssen, damit sie zusammenarbeiten können. Diese sind stark genug, so dass die Wissenschaftler die Interaktionspartner aus einem Proteinextrakt herausfischen können. «Im Fall der Komplexe rund um PP2A angelten wir gezielt 14 bekannte Proteine mit den daran gebundenen Proteinkomplexen heraus», erklärt Herzog. Die Wissenschaftler konnten zwar sagen, «wer mit wem» eine Bindung eingeht - unklar blieb, wo und wie die Proteine aneinander andocken.
Um diese Fragen zu beantworten, kombinierten die Wissenschaftler mehrere Analyseschritte. Zunächst bedienten sie sich eines Tricks: Mithilfe einer chemischen Substanz verbanden sie die zusammen herausgepflückten Proteinen an bestimmten Stellen fest miteinander. Und zwar immer dort, wo sich zwei bestimmte Aminosäuren (zwei Lysine) sehr nahe kamen. Dieses Verfahren heisst Cross-Linking, also die chemische Quervernetzung.
Danach zerlegten die Wissenschaftler die mit Quervernetzungen verbundenen Proteine in kleine Stücke und analysierten sie im Massenspektrometer. Mit einem ausgeklügelten Computerprogramm berechneten sie anschliessend, wo sich in den interagierenden Proteinen die Quervernetzungen befinden. Diese liegen dort, wo die beiden Proteine physisch in Kontakt treten.
Die Quervernetzungen liefern Information über den räumlichen Aufbau der einzelnen Proteinkomplexe. Sie ermöglichen zudem, die Kontaktflächen zwischen den Proteinen sowie die für die Bindung verantwortlichen Aminosäuren einzugrenzen. Die Forscher zeigten, dass es bei der Bindung, die PP2A mit dem Protein IGBP1 eingeht, 237 theoretische Möglichkeiten gibt, wie diese zueinander orientiert und aneinander angelagert sein könnten. Nach der Auswertung der Quervernetzung blieben nur noch vier sehr ähnliche räumliche Anordnungen der beiden Partner übrig.
Vielfältige Anwendungsmöglichkeiten
«Diese neue massenspektrometrischen Methode zur Strukturaufklärung ist hochinteressant für die Erforschung grosser Proteinkomplexe, die essenzielle Prozesse in der Zelle steuern und mit den herkömmlichen Methoden bisher nicht analysiert werden konnten», sagt Herzog.
Er selbst hat unlängst eine neue Stelle im Genzentrum an der Ludwig-Maximilians-Universität in München angetreten, wo er als Gruppenleiter die Methode auf Fragestellungen der Zellteilung anwenden wird. In der Gruppe von Ruedi Aebersold wird die Methode indes noch weiterentwickelt werden. Das Fernziel ist, sie so zu verfeinern, dass die Forscher die Proteinkomplexe ohne vorgängige Aufreinigung vielleicht sogar in lebenden Zellen quervernetzen können. Dadurch könnten sie erstmals ihre Strukturen und Interaktionen in ihrer «natürlichen» Umgebung genauer analysieren.
Literaturhinweis
Herzog F, Kahraman A, Böhringer D, Mak R, Bracher A, Walzthoeni T, Leitner A, Beck M, Hartl F-U, Ban N, Malmström L, Aebersold R. Structural Probing of a Protein Phosphatase 2A Network by Chemical Cross-Linking and Mass Spectrometry. Science, Online Publication 13th September 2012. DOI: 10.1126/science.1221483








LESERKOMMENTARE