Profiler auf Zellebene
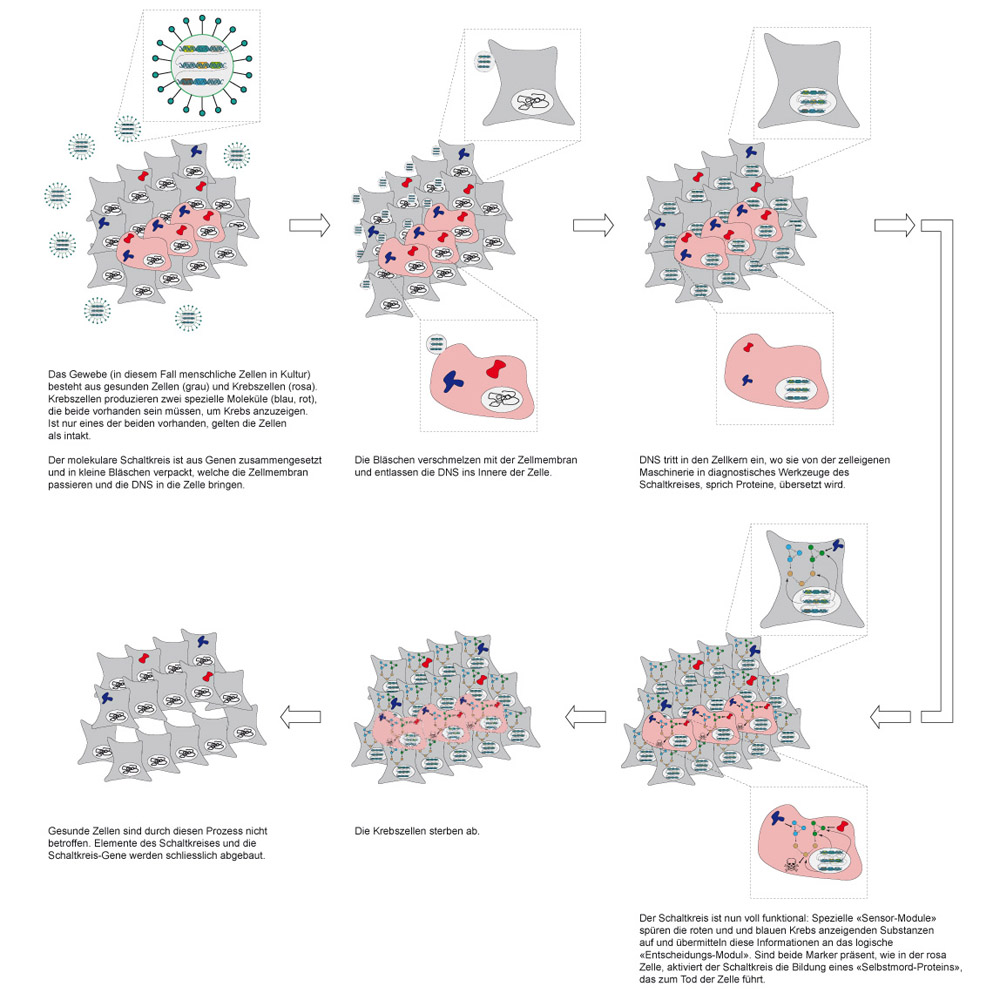
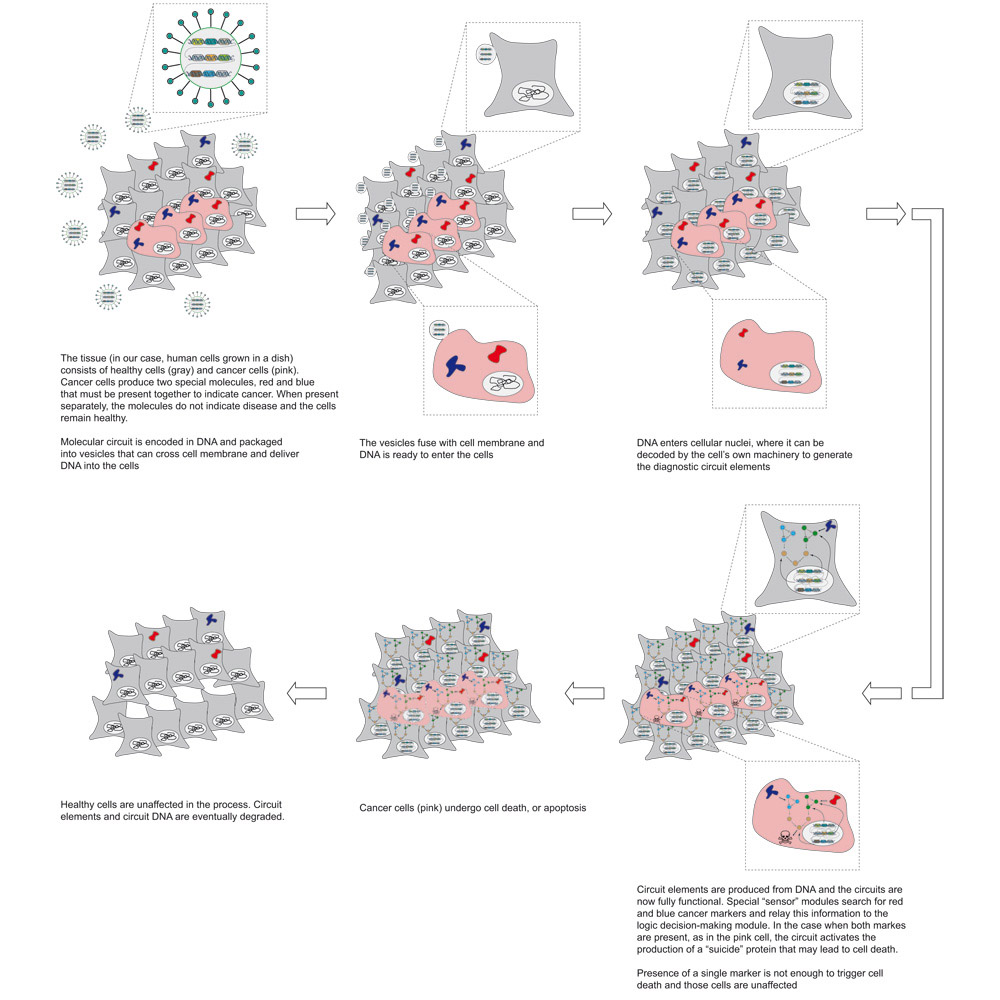
Forschern um ETH-Professor Yaakov Benenson und MIT-Professor Ron Weiss ist es gelungen, ein biologisches Computernetzwerk in menschliche Zellen einzubauen. Dieses Netzwerk erkennt spezifische Krebszellen anhand einer logischen Rechenoperation mit fünf Krebs-spezifischen Faktoren. Das führt zur Zerstörung der Krebszellen.
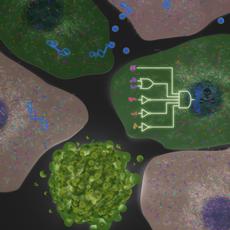
Schon sein ganzes Forscherleben lang arbeitet Kobi Benenson, Professor für Synthetische Biologie am D-BSSE, an biologischen Computern, die in lebenden Zellen arbeiten. Sein Ziel ist es, dass diese Biorechner im Inneren einer menschlichen Körperzelle verschiedene Moleküle erkennen, die wichtige Informationen über den Gesundheitszustand der Zelle preisgeben und entsprechend den gewonnenen Informationen eine Behandlung einleiten. Nun sind er und MIT-Professor Ron Weiss mit einem Team von Wissenschaftlern, den Postdoktoranden Zhen Xie und Liliana Wroblewska sowie Doktorandin Laura Prochazka, diesem Ziel einen grossen Schritt näher gekommen. In einer Publikation, die soeben in «Science» erschienen ist, stellen sie einen Schaltkreis aus verschiedenen Genen vor. Dessen Aufgabe ist es, zwischen Krebszellen und gesunden Zellen zu unterscheiden und richtig – sprich mit Zelltod der entarteten Zellen – zu reagieren. Das Netzwerk erkennt im Inneren der Zelle fünf Krebs-spezifische molekulare Faktoren sowie deren Konzentration. Weil der Schaltkreis nur funktioniert, wenn alle Faktoren in der Zelle vorhanden sind, ist die Identifizierung der Zelle sehr spezifisch
Krebszellen selektiv in den Tod geschickt
Die Forscher testeten das Gen-Netzwerk in Kulturen menschlicher Zellen: Krebszellen aus dem Gebärmutterhals, den sogenannten HeLa-Zellen, und gesunden Zellen. Diese Experimente waren erfolgreich. Nachdem die Forscher diesen genetischen Biorechner der Zellkultur beigegeben hatten, starben die HeLa-Zellen ab. Die gesunden Zellen hingegen blieben vom Zelltod verschont.
Für diesen Erfolg war viel Vorarbeit nötig. Benenson und sein Team mussten erst herausfinden, welche Kombination von Molekülen einzigartig für die HeLa-Zellen sind. Als Signalmoleküle dienten verschiedene Arten von Mikro-RNS (miRNS). Die Forscher mussten erst ein miRNS-Profil identifizieren, das für eine HeLa-Zelle typisch ist.
Das ist nicht einfach. Im Körper gibt es rund 250 verschiedene Zelltypen und unzählige Varianten von Krebszellen, wovon hunderte im Labor gezüchtet werden können. Noch grösser ist die Vielfalt von miRNS: 500 bis 1000 verschiedene Arten sind aus menschlichen Zellen bekannt. «Jeder Zelltyp, unabhängig davon ob gesund oder krank, hat verschiedene miRNS-Moleküle, die an- oder abgeschaltet sind», sagt Benenson.
Fünf Faktoren für Krebsprofil
Ein miRNS-Profil zu erstellen, ist nicht unähnlich einer Krankheitsdiagnose: «Ein Merkmal allein, wie beispielsweise Fieber, kann eine Krankheit nicht zuverlässig bestimmen. Je mehr Faktoren ein Arzt kennt, desto zuverlässiger wird seine Diagnose», erklärt der Professor, der vor eineinhalb Jahren von Harvard an die ETH gekommen ist. Die Forscher seines Teams haben deshalb auch nach mehreren Faktoren gesucht, die Krebszellen zuverlässig von gesunden Körperzellen unterscheiden.
In ihrem Versuch mit den HeLa-Zellen konnten die Wissenschaftler schliesslich fünf miRNSs identifizieren, die in einer bestimmten Konzentrationen vorliegen mussten, damit das Gen-Netzwerk die Zelle präzise und zuverlässig als Krebszelle identifizieren kann.
Operationen wie in einem Rechner
«Die Faktoren sind eigentlich Bool’sche Rechenoperationen. Jeder muss mit einem ‚AND‘- oder einem AND NOT-Befehl mit dem nächsten logisch verknüpft sein, damit am Schluss das richtige Resultat erzielt werden kann», erklärt Benenson.
Damit konnten die Forscher erstmals an lebenden Zellen demonstrieren, dass ihr Netzwerk sehr zuverlässig arbeitet. Dass der Zellcomputer gleich fünf verschiedene Faktoren miteinander verknüpfte und daraus die richtige Diagnose stellen konnte, ist für Benenson ein grosser Erfolg und ein wichtiger Schritt.
In einem nächsten Schritt will er diese Zellcomputer in einem geeigneten Tiermodell testen. Zukünftige Anwendungen könnten bei Diagnose und Therapie liegen. Das klingt zwar weiterhin nach Science Fiction, ist aber Benensons Ansicht nach machbar. Allerdings gibt es noch ein paar schwierige Probleme zu lösen, wie etwa die fremden Gene in einer Zelle effizient und sicher zu halten. Die Gene in die Zellen zu bringen ist ebenfalls nicht einfach. Für seinen Ansatz verabreicht der ETH-Professor temporär Gene mit Viren oder chemischer Verbindungen. Diese Methoden sind jedoch nicht ausgereift und sollten zukünftig verbessert werden.
«Von einer voll funktionalen Behandlungsmethode für Menschen sind wir noch immer sehr weit entfernt. Diese Arbeit aber ist ein erster, wichtiger Schritt, der die Machbarkeit solch selektiver Diagnosemethode aufzeigt», sagt Benenson.
Literaturhinweis
Xie Z, Wroblewska L, Prochazka L, Weiss R, Benenson Y. Multi-Input RNAi-Based Logic Circuit for Identification of Specific Cancer Cells. Science, published online 1st September 2011. doi:10.1126/science.1205527








LESERKOMMENTARE