Fischen nach Wirkstoffen
Mit einer neuen Screening-Methode, die auf einer DNS-kodierten Chemikalien-Sammlung basiert, versuchen ETH Forscher der Arbeitsgruppe von Dario Neri, Professor für Biomakromoleküle am Institut für Pharmazeutische Wissenschaften, die Suche nach potenziellen Wirkstoffen für Medikamente zu revolutionieren. Die Methode ist einfach, schnell und günstig. Im Rahmen dieses Projektes forscht die Arbeitsgruppe bereits nach neuen Wirkstoffen gegen rheumatoide Arthritis.
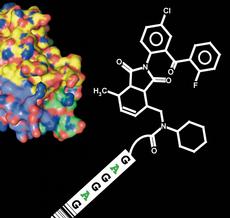
Dario Neri, Professor am Institut für Pharmazeutische Wissenschaften der ETH Zürich, spricht von einer möglichen Revolution des Screenings von Wirkstoffen, die die neue, in seinem Labor entwickelte Methode einer «DNA-encoded Chemical Library» auslösen kann. Dabei handelt es sich um eine Sammlung von unzähligen verschiedenen kleinen Molekülen, an welche ein kurzes Stück DNS als individuelle Erkennungsmarke geknüpft wurde, ähnlich dem Barcode eines Produkts.
Für die neue Methode, an deren Entwicklung Neris Doktorand Fabian Buller massgeblich beteiligt ist, , finanziell unterstützt von der Förderagentur für Innovation KTI und der Gebert Rüf Stiftung, dürfte sich auch die Pharmaindustrie interessieren: Denn Verfahren zum Durchtesten von möglichen Wirkstoffen sind bis heute zeitaufwändig und sehr teuer. Sobald ein krankheitsrelevantes Zielprotein entdeckt ist, werden in der Pharmaindustrie Substanzbibliotheken, die etwa eine Million Substanzen enthalten, mit Robotersystemen durchgetestet. Auch die Weiterverfolgung der «Treffer» ist oft so aufwändig, dass in erster Linie nur grosse Pharmaunternehmen diese Methode anwenden können.
Einfacher, schneller, billiger
Die Methode, die Fabian Buller und andere Wissenschaftler in der Fachzeitschrift «Chemistry & Biology» vorgestellt haben, vereinfacht und miniaturisiert das Wirkstoff-Screening. Ihre Substanzenbibliothek kann eine Million bis eine Milliarde verschiedener Substanzen enthalten und hat Platz in einem sogenannten Eppendorf-Gefäss, einem Mini-Reaktionsgefäss von etwa einem halben Milliliter Inhalt. Damit kann zum ersten Mal eine grosse Zahl von kleinen Molekülen rasch und kostengünstig auf potenzielle Kandidaten für Medikamente getestet werden. Die kleinen organischen Binder liegen in der Bibliothek nur in extrem kleiner Konzentration vor, so dass schon kleinste Mengen des Zielproteins reichen, um die gesamte Substanz-Sammlung auf Wechselwirkungen zu testen.
Um potenzielle Wirkstoffe zu finden, mischen die Forscher in einem einzigen Experiment ihre Substanz-Bibliothek mit einem immobilisierten Zielprotein. Moleküle, die nicht an dieses binden, lassen sich wegwaschen, die «Treffer» jedoch können die Forscher anhand des DNS-Codes rasch und eindeutig mittels einer Polymerase-Kettenreaktion (PCR), mit der die DNS vervielfältigt wird, und anschliessender High-throughput Sequenzierung identifizieren.
Treffer aus 4000 Substanzen
Um die Methode zu testen, erstellte Buller in einem ersten Schritt eine Sammlung von 4000 kleinen Molekülen. Diese wurden auf Wechselwirkung mit dem Tumornekrosefaktor (TNF) untersucht. TNF ist ein Signalstoff des Immunsystems, der bei Entzündungen gebildet wird, zum Beispiel auch bei rheumatoider Arthritis. Für diese Krankheit gibt es mehrere Medikamente auf dem Markt, die TNF blockieren und Milliardenumsätze erzielen. Diese Medikamente basieren aber ausschliesslich auf Antikörpern und nicht auf kleinen organischen Molekülen.
Aus den 4000 Substanzen konnten die Forscher einige Treffer finden, die mit dem TNF-Proteinkomplex in Wechselwirkung traten. Nach chemischer Verfeinerung testeten sie überdies den besten Kandidaten in einem Zellkulturexperiment. Die gefundene Substanz erlaubte den Zellen zu überleben, in dem sie das TNF-Signal blockierte. Zum ersten Mal wurde damit gezeigt, dass es prinzipiell möglich sein sollte, einen TNF-Blocker auf der Basis eines kleinen Moleküls zu entwickeln.
Bisherige TNF-Blocker mit Nachteilen
Denn die bisherigen auf Antikörpern basierten Medikamente haben Nachteile: Sie können immunogen sein, also im Körper nach mehrmaliger Anwendung eine Immunantwort auslösen. In diesem Fall können sie nur über kurze Zeit verabreicht werden, das Medikament wirkt dann nicht mehr und die Patienten müssen auf ein anderes Medikament wechseln. Diese Medikamente müssen zudem mit einer Spritze injiziert werden. Der Vorteil eines TNF-Blockers auf Basis eines kleinen Moleküls wäre, dass dieses nicht immunogen und auch patientenfreundlicher wäre, da das Medikament oral verabreicht werden könnte.
Die mit dem neuen Screening aus einem Meer von Kandidaten «gefischten» Moleküle sind zwar noch keine fertigen Wirkstoffe und müssen, ehe sie zum Einsatz kommen, verbessert und klinisch getestet werden. Das Beispiel aber zeigt, dass die vergleichsweise einfache neue Screening-Methode rasch vielversprechende Resultate liefert.
Darüber hinaus kann die Substanz-Sammlung rasch vergrössert werden. Die ETH-Forscher haben ihre DNS-kodierte Molekülbibliothek bereits auf eine Million Substanzen erweitert. In Zukunft will die Arbeitsgruppe von Dario Neri diese sogar auf mehr als 100 Millionen verschiedene kleine Moleküle ausbauen. Auf diese Weise wollen die Wissenschaftler das Wirkstoffscreening optimieren und auf schnelle und kostengünstige Weise kleine Moleküle gegen klinisch relevante Zielproteine entwickeln - und so auch neue Medikamente gegen rheumatoide Arthritis finden.
Literaturhinweis:
Buller F et al. Discovery of TNF Inhibitors from a DNA-Encoded Chemical Library based on Diels-Alder Cycloaddition. Chemistry & Biology, Volume 16, Issue 10, 1075-1086, 30 October 2009. doi:10.1016/j.chembiol.2009.09.011








LESERKOMMENTARE